以前、仮想環境の構築やバイオデータベースのファイルをご紹介しました。今度はこちらを活用し、実際に解析を行ってみましょう!
バイオインフォマティクスの解析は、主に「コマンド」を用いて行われています。以下のフローでよく使うコマンドを用いて、解析してみます。
- 1. ターミナルを開く
-
コマンドはUbuntuでは「ターミナル」で打ち込むことができます。起動方法はいくつかパターンがあります。
- 画面から起動 : [アプリケーション] → [アクセサリ] → [端末]
- ショートカットキーで起動 : Ctrl + Alt + T
- 2. ディレクトリを移動する
-
「ディレクトリ」とはWindowsで言うフォルダと同意です。ターミナルを起動した時にいる場所は、ホームディレクトリと言われる場所です。
まずは解析したいファイルの場所まで移動します。この時使用するのは「cd」コマンドです。解析したいファイルがデスクトップにある場合、以下のように打ち込みEnterを押します。「cd /home/{ユーザー名}/Desktop」
({ユーザー名}の部分はそれぞれログインしているユーザー名を入れます) - 3. ファイルを確認する
-
移動ができたら、今度はファイルの存在を確認してみます。移動したディレクトリにあるファイルのリストを表示するには「ls」コマンドを使用します。こちらのコマンドは「ls」と打ち込みEnterを押すだけでOKです。

- 4. 解析してみる
-
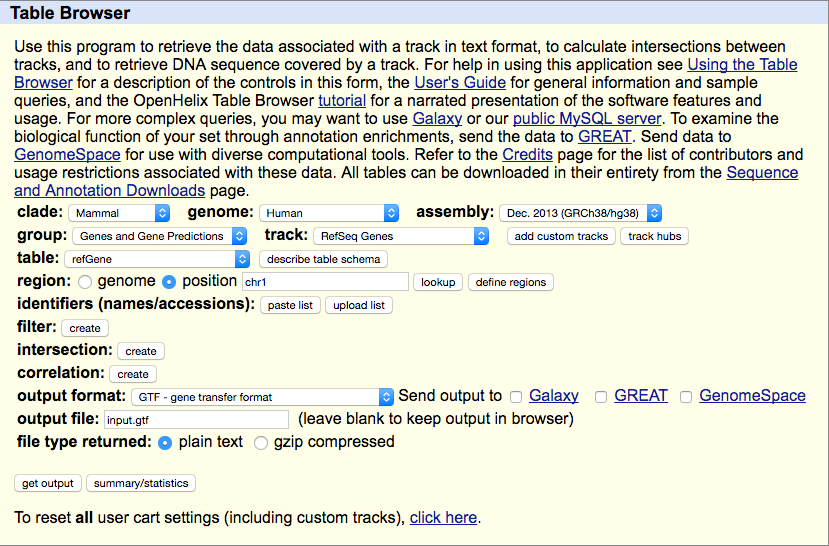
ここではUCSCから取得したデータを解析してみることにいたします。Table Browserより、以下の画像のパラメーターを設定してGTF形式ファイルを取得しました。
取得したGTFファイルになんの情報が入っているのか、「feature」から確認しましょう。
- feature 情報の取り出し
-
GTFファイルはタブ文字区切りで結果が書かれております。「cut」コマンドで3区切りめの feature を取り出して保存するには以下のように打ち込みEnterを押します。
「cut -f 3 input.gtf > feature.txt」
「-f」はオプションと呼ばれるもので、数字を指定することで区切り位置を決定しています。
cutコマンドの区切り文字はデフォルトでタブ文字が指定されています。
「>」は出力先を指定しており、この場合 feature.txt ファイルを作成し保存しております。 - ファイルの中身を確認
-
正しく feature が取り出せたか、ファイルの中身を確認するには「cat」コマンドです…が、今回はレコード数が多いデータですので、先頭のみを確認する「head」コマンドを使用します。
「head feature.txt」
「-n」オプションに数値を指定することで、先頭行からみたい行数まで表示される様になります。
デフォルトは10行になっています。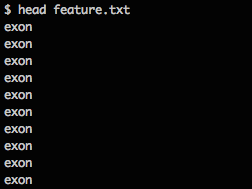
- featureから重複を削除する
-
GTFのfeatureは種類ごとにまとまっていません。重複を削除にするには2段階コマンドが必要になります。
1段階めは「sort」コマンドで行を並び替えます。「sort feature.txt > sort.txt」2段階めに「uniq」コマンドで重複削除です。
「uniq sort.txt > uniq.txt」
「uniq」コマンドは上下の行が重複している場合、まとめる効果があるため、まずはソートする必要があります。結果はまた「cat」コマンドで確認します。
「cat uniq.txt」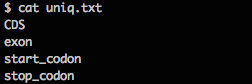
いかがでしたでしょうか?ここで紹介した解析は、実はエクセルでもできます。しかし巨大なファイルはエクセルでは開けないことがあります。データベースから取得したファイルをUbuntuで解析してみてはいかがでしょうか?