The Cancer Genome Atlas(TCGA)では膨大ながん関連遺伝子発現データや腫瘍画像データが公開されており、日々がん研究に役立てられています。
今回は、The Cancer Genome AtlasからRNA-seqの発現量データをLinuxコマンドで一括ダウンロードする方法を説明します。
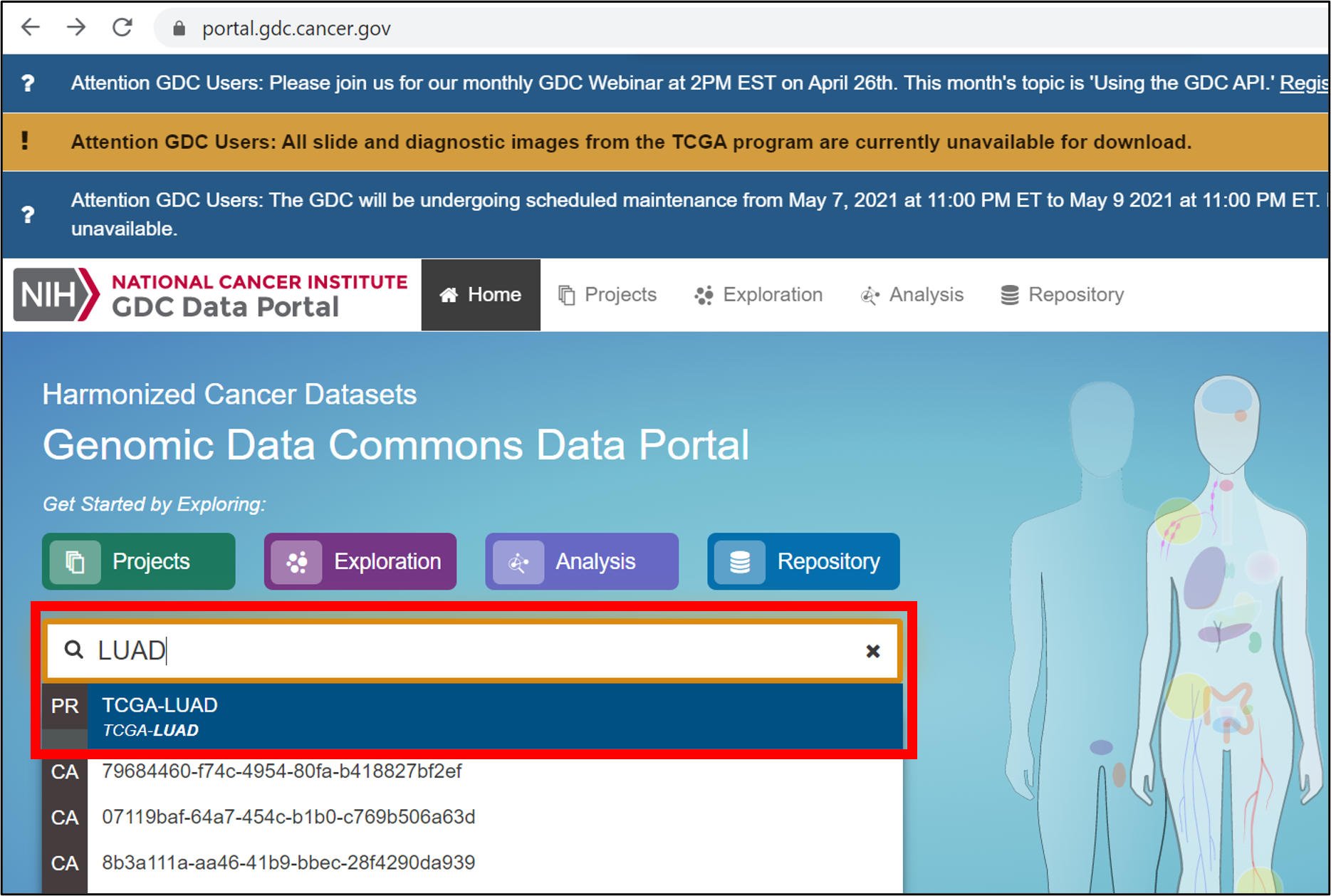
まずはThe Cancer Genome Atlasのサイトにアクセスして、下段にある「Access TCGA Data」から 検索ページに飛びます。
 図1、The Cancer Genome Atlasのトップページ
図1、The Cancer Genome Atlasのトップページ
検索窓に「LUAD」(=Lung Adenocarcinoma、肺腺癌)と入力すると、登録されているLUAD関連サンプルの候補が表示されます。今回はTCGA-LUADを選択して検索します。
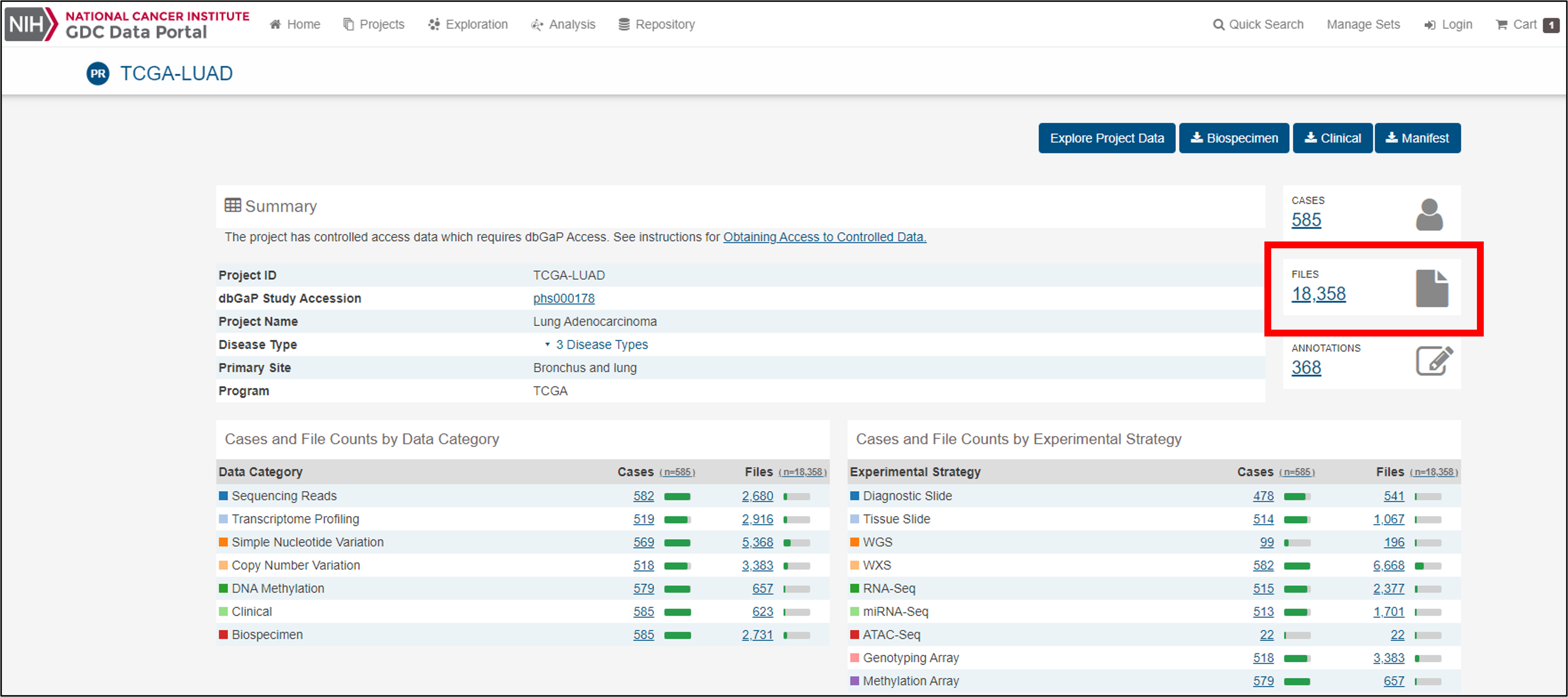
検索するとTCGA-LUAD関連データ一覧が表示されるので、このうち右側の「Files」をクリックします。
図3、TCGA-LUAD関連データ一覧と、Filesボタン
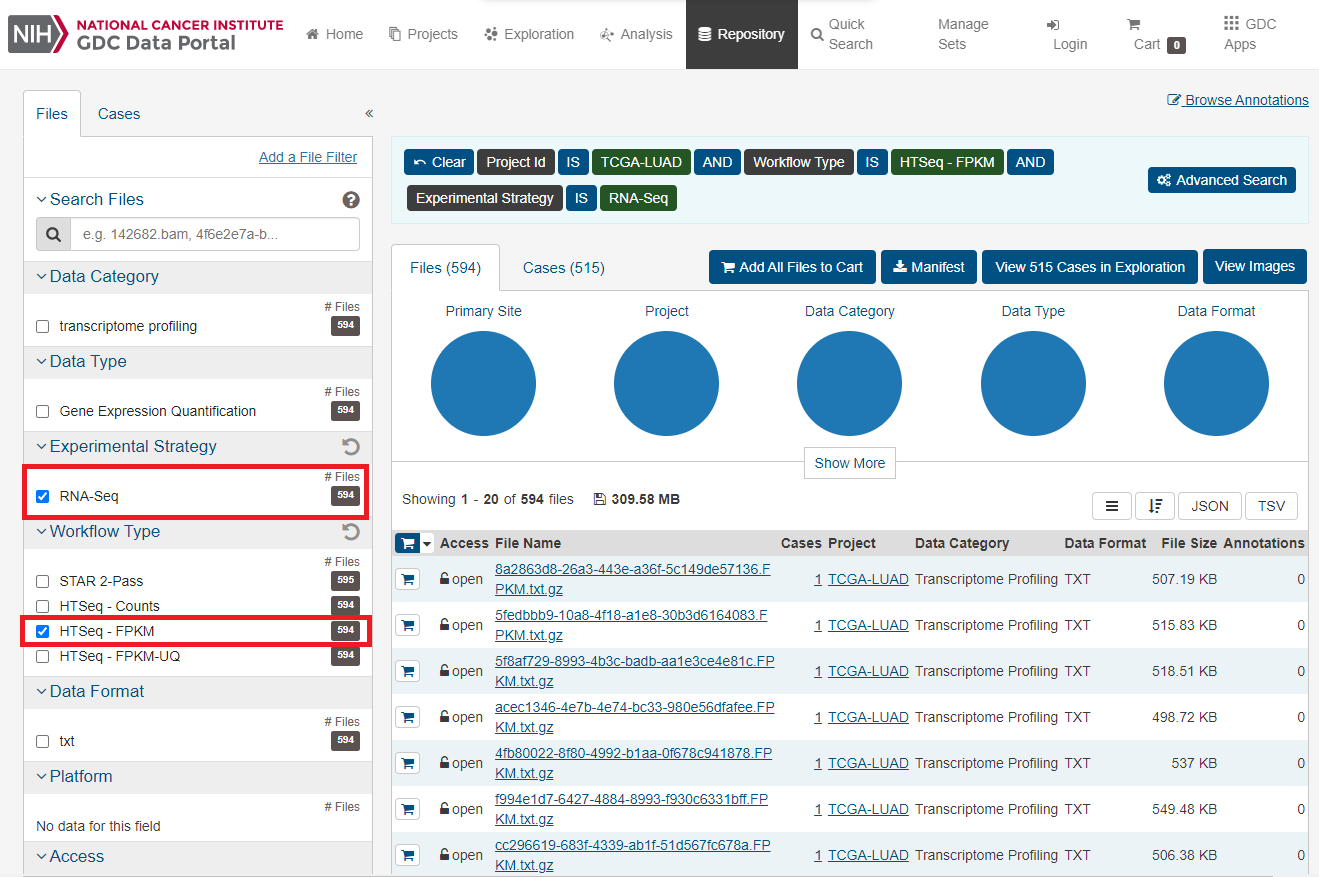
左側のメニューにおいて「Experimental Strategy」で「RNA-seq」、「Workflow Type」で「FPKM」にチェックを入れると、RNA-seqの補正済み発現量カウントデータ(FPKM)のみに表示が自動で切り替わります。
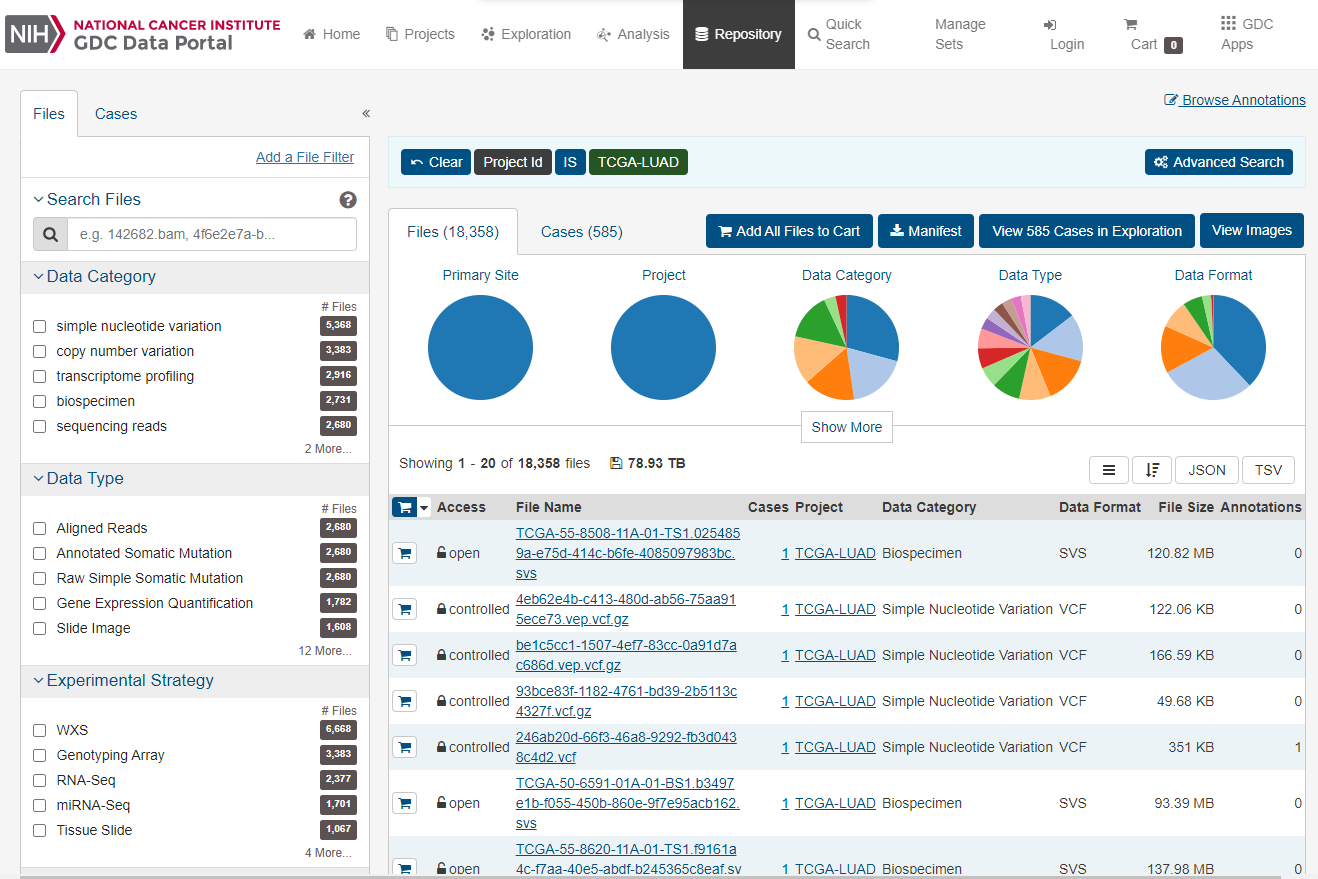
図4、項目選択前
図5、「RNA-Seq」と「FPKM」を選択後の画面
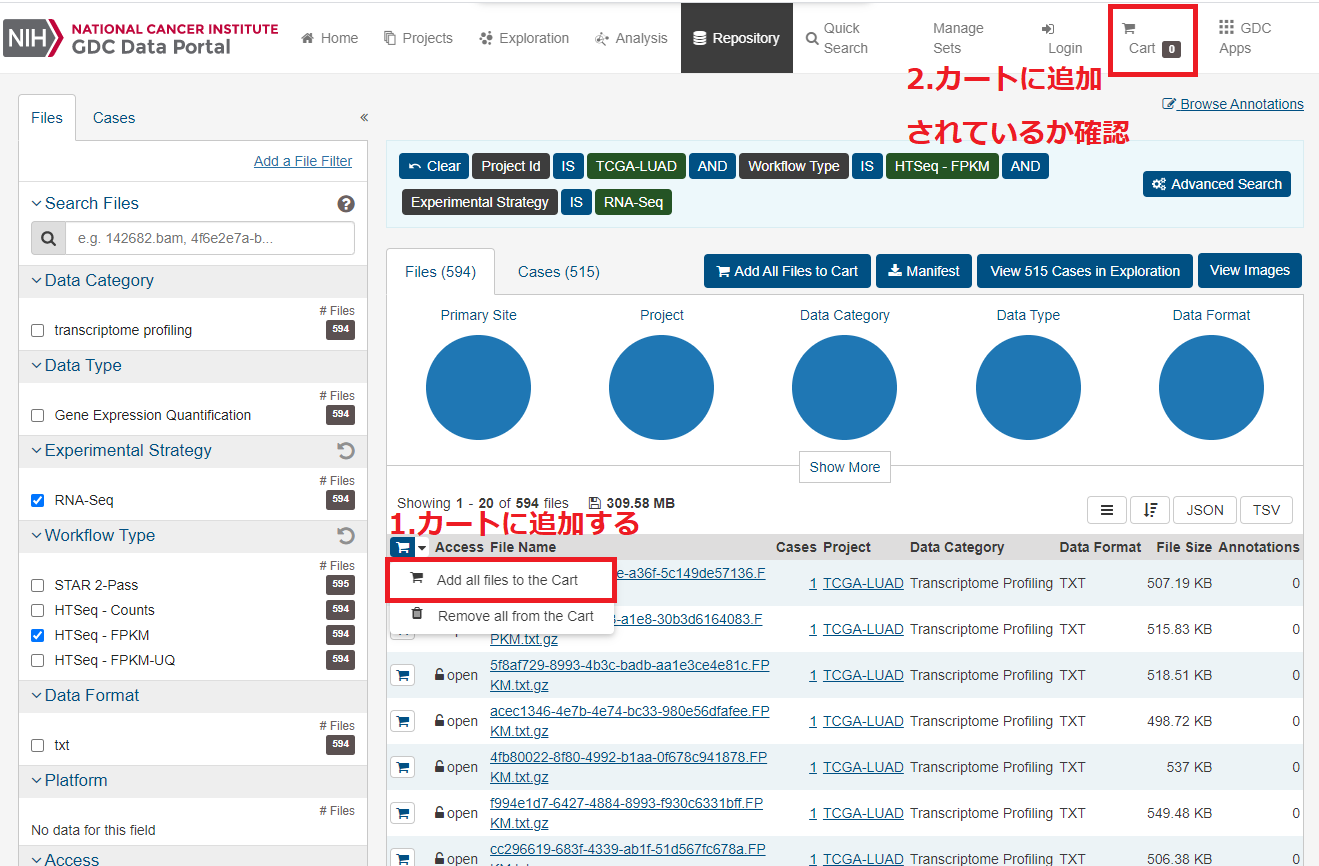
選択されているファイルをダウンロードするためには「Add all files to Cart」ボタンを押し、画面右上のCartに追加されることを確認します。
図6、カートに追加する画面
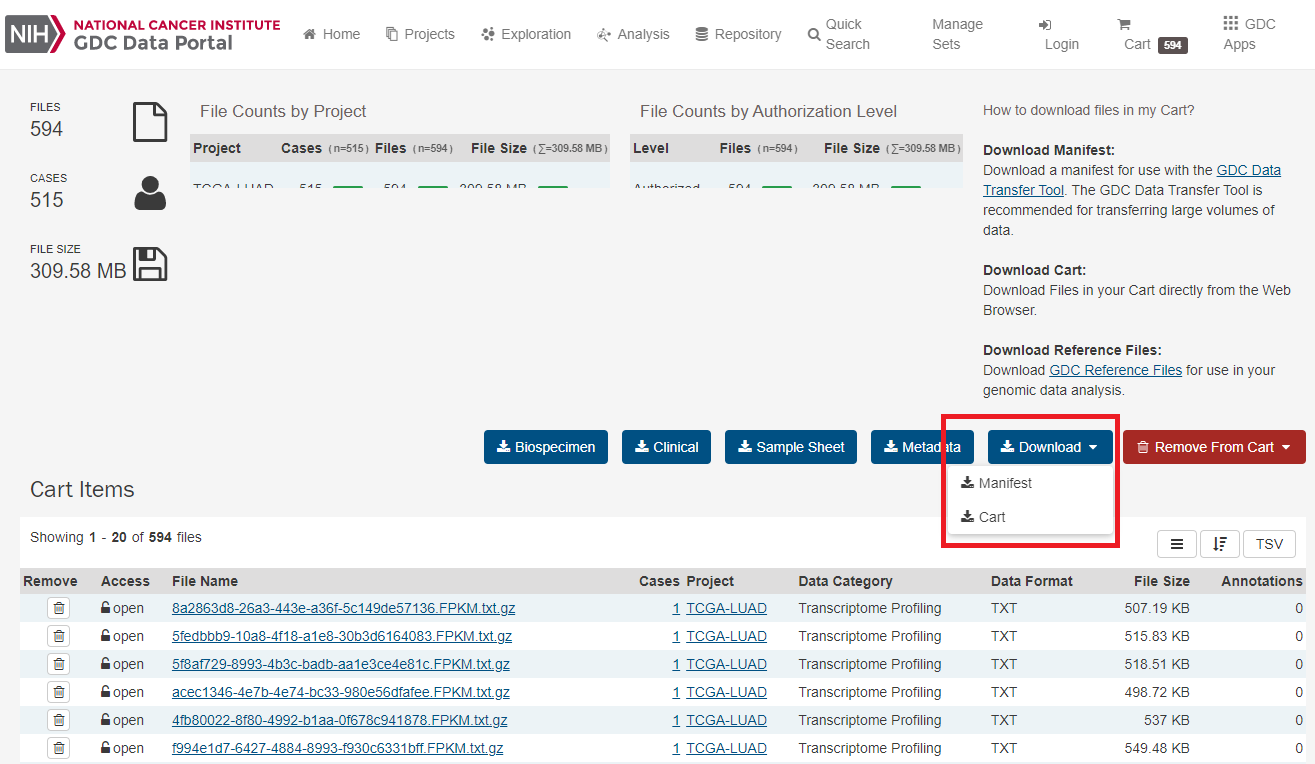
右上のカートボタンを押して、カートの中身を確認します。
図7、カートの中身とダウンロードボタン
カートにある「Download」→「Cart」ボタンから直接ダウンロードすることも可能です。今回はファイル数が多いので、手元のPCではなく直接Linuxサーバー(CentOSなど)に一括ダウンロードすることを考えます。
まずは図7にある「Download」→「Manifest」ボタンからManifestファイルをダウンロードしておきます。今回はダウンロードしたManifestファイル名を「gdc_sample_sheet_LUAD_htseq_fpkm.tsv」とします。このファイルの情報に基づいて後のダウンロードが行われるので、このファイルをLinuxサーバー側にscpコマンドなどで転送しておきます。
次にLinuxサーバーにgdc-clientツールをダウンロードします。
https://gdc.cancer.gov/access-data/gdc-data-transfer-tool
にアクセスし、下段にある Linux用Binary をダウンロードします。
[CentOS]$ wget https://gdc.cancer.gov/files/public/file/gdc-client_v1.6.1_Ubuntu_x64.zip
ダウンロードしたzipフォルダーを解凍するとgdc-clientができるので、実行権限が無ければchmodで実行権限を与えます。
[CentOS]$ unzip gdc-client_v1.6.1_Ubuntu_x64.zip
[CentOS]$ chmod +x gdc-client
gdb-clientに先ほど転送しておいたManifestファイル-mで指定すればダウンロードが開始されます。-dで指定されるダウンロード先フォルダーはmkdirコマンドで事前に作成しておく必要があります。
[CentOS]$ ./gdb-client download -m gdc_sample_sheet_LUAD_htseq_fpkm.tsv -d download_dir/
データが大きくダウンロードには時間がかかるので、nohup [コマンド] & を付けて実行することが推奨されます。
[CentOS]$ nohup ./gdc-client download -m gdc_sample_sheet_LUAD_htseq_fpkm.tsv -d download_dir/ &